올해의 CRISPR/Cas 시스템 노벨상은 1990년대 가이드 RNA(gRNA)의 획기적인 발견으로 시작된 중요한 과학적 여정을 조명합니다. 이 기초 연구는 주요 과학적 돌파구가 종종 이전의 발견 위에 구축되는 방식을 강조합니다.
주제 분류: 과학사 및 철학, RNA 생물학
CRISPR-Cas9 시스템은 분자 생물학에서 없어서는 안 될 존재가 되었습니다. 자연에서 이 시스템은 박테리아의 적응 면역 시스템 역할을 하여 과거 바이러스 감염을 기억하고 방어할 수 있도록 합니다. 그러나 CRISPR-Cas9는 놀랍도록 다재다능하고 정밀한 유전자 편집 도구로 가장 유명합니다. 이 혁신적인 응용 프로그램은 Emmanuelle Charpentier와 Jennifer Doudna에게 2020년 노벨 화학상을 안겨주었습니다(Jinek, Chylinski et al, 2012). 이 시스템의 핵심은 특정 DNA 서열을 표적으로 삼아 이중 가닥 절단을 생성하는 프로그래밍 가능한 엔도뉴클레아제인 Cas9이며, 이는 다양한 유전자 변형 기술에 활용될 수 있습니다. Cas9의 특이성은 가이드 RNA(gRNA)로 알려진 작은 RNA 분자에 의해 결정됩니다. 이 gRNA는 표적 DNA 서열과 염기쌍을 이루고 동시에 Cas9와 상호 작용하여 부위 특이적 DNA 절단을 시작합니다(그림 1). 흥미롭게도 첫 번째 가이드 RNA의 발견과 특정 핵산 서열에서 RNA 지시 단백질 활성의 기본 원리는 CRISPR-Cas9 혁명보다 20년 이상 앞서 있습니다.
그림 1. “가이드 RNA” 및 다양한 역할에 대한 연대순 개요.
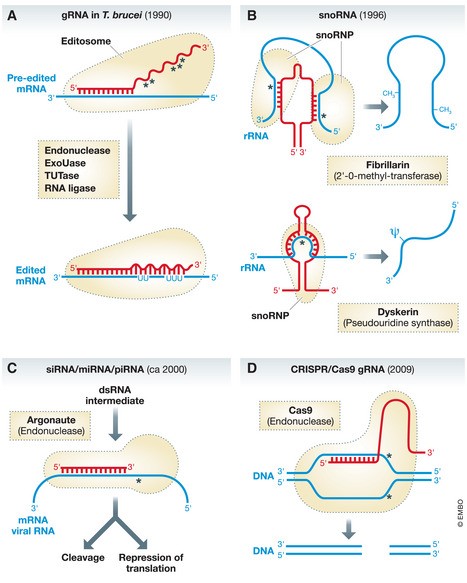 가이드 RNA의 종류별 비교: 트리파노소마 미토콘드리아 RNA 편집, snoRNA에 의한 rRNA 변형, siRNA/miRNA 유전자 조절, CRISPR/Cas9 DNA 표적
가이드 RNA의 종류별 비교: 트리파노소마 미토콘드리아 RNA 편집, snoRNA에 의한 rRNA 변형, siRNA/miRNA 유전자 조절, CRISPR/Cas9 DNA 표적
(A) 트리파노소마 및 미토콘드리아 RNA 편집의 가이드 RNA: 이러한 gRNA는 미토콘드리아 내에서 사전 편집된 RNA의 우리딘 삽입 및 삭제를 지시합니다. 이 과정에는 엔도리보뉴클레아제, 말단 우리딜트랜스퍼라제(Tutase), U 특이적 엑소리보뉴클레아제(ExoUase) 및 RNA 리가아제와 같은 일련의 효소 활성이 포함됩니다. 빨간색 물결선은 RNA 편집을 안내하는 gRNA 영역을 나타냅니다. (B) rRNA 변형의 snoRNA: 작은 핵소체 RNA(snoRNA)는 두 가지 유형의 리보솜 RNA(rRNA) 변형(2′-O-메틸화 및 슈도우리딜화)을 안내합니다. 각 변형 유형에는 서로 다른 snoRNA 세트가 담당합니다. (C) 유전자 조절의 siRNA 및 miRNA: 작은 간섭 RNA(siRNA)와 마이크로RNA(miRNA)는 서로 다른 경로를 통해 처리되어 이중 가닥 RNA 중간체를 생성합니다. 이러한 중간체의 센스 가닥은 Argonaute 계열 단백질을 포함하는 단백질 복합체에 통합됩니다. siRNA와 miRNA는 표적 RNA 분해를 유도하거나 번역 및 전사를 조절하여 유전자 발현을 조절합니다. (D) 표적 DNA 절단을 위한 CRISPR/Cas9 시스템: CRISPR/Cas9 시스템은 가이드 RNA(gRNA)를 사용하여 Cas9 엔도뉴클레아제를 특정 DNA 서열로 유도하여 이중 가닥 절단을 생성합니다. 이 시스템은 다양한 유기체에서 유전 공학에 혁명을 일으켰습니다. 여기에 표시된 최적화된 시스템은 gRNA와 tracRNA의 융합을 특징으로 합니다. 원래의 원핵생물 CRISPR-Cas 시스템에서 Cas9는 개별 gRNA 및 tracRNA 분자와 결합하여 바이러스 감염에 대한 적응 면역을 제공합니다. 빨간색 영역은 “가이드 RNA” 세그먼트를 나타냅니다. 파란색은 “gRNA” 표적 서열을 나타냅니다. 끊어진 검은색 선은 “gRNA”와 관련된 단백질 또는 단백질 복합체를 나타냅니다. gRNA와 표적 핵산 사이의 염기쌍 영역이 강조 표시됩니다. 별표는 표적 핵산의 절단 또는 변형 부위를 표시합니다.
… 최초의 gRNA의 발견 […]은 CRISPR/Cas9 시스템의 발견보다 20년 이상 앞서 있습니다.
이 기사는 “가이드 RNA”라는 용어의 역사를 탐구하여 그 기원과 개념의 발전을 추적합니다. 이러한 초기 발견에 직접 관여하지는 않았지만 저자는 특히 1990년 그의 기념비적인 논문에서 처음으로 gRNA라는 용어를 만들어낸 Beat Blum에 대한 개인적인 통찰력을 공유합니다(Blum et al, 1990). Beat Blum의 gRNA 발견은 과학적 돌파구로 이어지는 종종 복잡하고 구불구불한 경로를 보여주는 설득력 있는 예이며, 그의 이야기를 여기에서 중심적으로 다룹니다.
RNA 편집 해독: 가이드 RNA의 기원
이야기는 암스테르담 대학교의 Rob Benne에서 시작됩니다. 그는 기생 원생동물인 Trypanosoma brucei의 미토콘드리아 DNA와 해당 mRNA를 조사하고 있었습니다. Benne의 시토크롬 산화효소 서브유닛 2(Cox2) 분석 결과 놀라운 차이가 드러났습니다. 게놈 DNA 서열과 mRNA 유래 서열이 동일하지 않았습니다. 그는 mRNA에서 유전자에는 없는 4-우리딘 삽입을 지속적으로 관찰했습니다. 이 삽입은 Cox2 유전자에서 예측된 프레임 이동을 수정했습니다. 처음에는 믿기 어려웠지만 1986년에 발표된 Benne의 연구 결과는 RNA 편집이라는 새로운 과정을 제안했습니다. 이 과정은 Cox2 전사체의 특정 위치에 4개의 우리딘을 정확하게 삽입하여 기능적이고 번역 가능한 mRNA를 생성했습니다(Benne et al, 1986).
처음에는 Benne의 논문에 대한 회의론이 있었지만, 트리파노소마티드 내에서 더 극적인 RNA 편집의 추가적인 예가 발견됨에 따라 이러한 회의론은 점차 사라졌습니다. 이러한 편집 이벤트에는 여러 개의 우리딘 삽입 및 삭제가 포함되었으며, 어떤 경우에는 성숙한 mRNA가 더 이상 해당 유전자와 혼성화되지 않을 정도로 광범위했습니다. RNA 편집은 과학계에서 주목을 받았고 그 의미에 대한 수많은 리뷰가 나왔습니다. 가장 궁금한 질문은 이러한 편집된 서열에 대한 정보의 출처였습니다. 광범위한 검색에도 불구하고 트리파노소마 미토콘드리아 게놈에서 편집된 서열과 일치하는 DNA 또는 RNA 주형을 찾을 수 없었습니다. 이로 인해 일부 사람들은 RNA 편집이 Francis Crick의 분자 생물학의 중심 원리를 뒤흔들 수 있다고 생각하게 되었고, 기본 메커니즘에 대한 비전통적인 이론을 촉발했습니다.
… 일부 사람들은 RNA 편집이 Francis Crick의 중심 원리를 뒤흔들 것이라고 생각했고, 이는 그 과정이 어떻게 작동할 수 있는지에 대한 몇 가지 매우 억지스러운 아이디어로 이어졌습니다.
최초의 가이드 RNA의 등장
이 기간 동안 Beat Blum과 저자는 베른 대학교의 Thomas Seebeck 연구실에서 박사 과정 학생으로 T. brucei를 연구했습니다. Beat은 새롭게 발견된 RNA 편집 현상에 매료되었습니다. 박사 학위를 마친 후 그는 이 분야의 선두 전문가인 UCLA의 Larry Simpson과 함께 박사후 연구원 과정을 밟았습니다. Beat은 RNA 편집을 안내하는 정보의 출처를 밝히겠다는 야심찬 제안과 함께 스위스 국립 과학 재단(SNF)에서 박사후 연구원 자격을 얻었습니다. 다른 사람들과 달리 Beat은 중심 원리를 고수했으며 해결책은 편집된 서열을 보완하는 핵산과의 특정 염기쌍에 있다고 가정했습니다. 그의 SNF 제안서는 이러한 가상의 핵산을 식별하기 위한 다양한 전략을 자세히 설명했습니다. RNA 편집 퍼즐의 이 누락된 조각을 찾는 것은 Beat이 Simpson의 연구실에 합류하기 전에도 그의 흔들리지 않는 임무가 되었습니다. 그는 귀를 기울이는 모든 사람에게, 그리고 그렇지 않은 많은 사람들에게 자신이 Larry Simpson의 연구실에 가서 RNA 편집의 미스터리를 완전히 해결할 것이라고 자신 있게 선언했습니다. 당시에는 저자를 포함한 많은 사람들이 회의적이었습니다.
… RNA 편집은 Beat의 임무가 되었고 그는 귀를 기울이는 모든 사람에게, 그리고 그렇지 않은 많은 사람들에게 자신이 [..] RNA 편집 문제를 완전히 해결할 것이라고 말했습니다.
그러나 Larry Simpson의 연구실에 도착한 직후 Beat은 중요한 돌파구를 마련했습니다. 그는 계산 분석을 수행하고 또 다른 트리파노소마티드인 Leishmania tarentolae의 미토콘드리아 게놈에서 편집된 mRNA의 세그먼트와 놀라울 정도로 유사한 짧은 서열을 식별했습니다. 이러한 서열은 또한 3′ 측면에서 동일한 mRNA의 편집되지 않은 영역과 밀접하게 일치하는 짧은 스트레치로 측면에 배치되었습니다. 다른 사람들이 사용 가능한 서열 데이터를 사용하여 유사한 접근 방식을 시도했지만 성공하지 못했습니다. Beat의 핵심 혁신은 그의 분석에서 구아닌-우라실(G-U) 염기쌍을 허용한 것입니다. G-U 염기쌍은 알려진 현상이었고 RNA 기능에서의 중요한 역할은 EMBO Reports 창간호에 게재된 리뷰에서 강조되었습니다(Varani & McClain, 2000).
Beat은 이후 편집된 영역과 일치하는 이러한 서열이 실제로 짧은 RNA로 전사되었음을 입증했습니다. 1990년 1월에 그는 Larry Simpson, Norbert Bakalara와 함께 이러한 연구 결과를 Cell에 발표했습니다(Blum et al, 1990). 과학 문헌에서 이 용어를 처음으로 문서화하여 “가이드 RNA”라는 용어를 도입했습니다. 그들은 gRNA의 5′ 부분이 사전 편집된 mRNA의 측면 영역과 혼성화되어 RNA 편집 기계를 정확한 편집 부위로 안내하는 모델을 제안했습니다. RNA 유도 효소 활성 표적화라는 이 개념은 이제 광범위하게 gRNA 개념으로 인식되는 것의 본질입니다.
트리파노소마 gRNA는 최초로 발견되었을 뿐만 아니라 현재까지 알려진 것 중 가장 복잡하다는 점에 유의해야 합니다. 그들은 엔도뉴클레아제, 말단 우리딜트랜스퍼라제, U 특이적 엑소리보뉴클레아제 및 RNA 리가아제 활성을 포함하는 RNA 편집 기계를 사전 편집된 mRNA의 올바른 위치로 조정합니다. 그런 다음 이러한 gRNA는 이러한 효소에 정확한 수의 우리딘을 순차적으로 삽입하거나 삭제하도록 지시합니다(그림 1). 오늘날 우리는 mRNA 코딩 서열의 전사 후 변형인 RNA 편집이 광범위하고 다양하다는 것을 알고 있습니다. 그러나 놀랍게도 트리파노소마티드 미토콘드리아의 RNA 편집은 가이드 RNA에 의존하는 것으로 알려진 유일한 사례로 남아 있습니다.
snoRNA: 또 다른 종류의 가이드 RNA
트리파노소마 gRNA가 발견된 지 6년 후, 또 다른 종류의 작은 RNA인 작은 핵소체 RNA(snoRNA)도 가이드 RNA로 기능하는 것으로 밝혀졌습니다. 핵소체에 존재하는 것으로 오랫동안 알려져 온 snoRNA는 Zsuzsanna Kiss-Laszlo에 의해 리보솜 RNA(rRNA)에서 특정 뉴클레오티드의 2′-O-메틸화를 안내하는 것으로 나타났습니다(Kiss-Laszlo, Henry et al, 1996). 1년 후 동일한 그룹은 서로 다른 snoRNA 세트가 rRNA에서 우리딘을 슈도우리딘으로 부위 특이적으로 전환시키는 것을 지시한다는 것을 입증했습니다. 진핵 세포에는 수백 개의 서로 다른 snoRNA가 포함되어 있으며, 그 중 다수는 인트론 내에 인코딩되어 있습니다. 이러한 snoRNA는 변형 효소를 포함한 관련 단백질과 함께 snoRNP라고 하는 리보핵단백질 입자(RNP)를 형성합니다. 각 snoRNA에는 rRNA 표적에서 변형될 뉴클레오티드를 둘러싼 영역을 보완하는 뉴클레오티드 서열이 포함되어 있습니다(그림 1). 이러한 상보성을 통해 snoRNA는 표적 RNA를 인식하고 변형 효소를 올바른 위치로 모집할 수 있습니다. 박테리아 rRNA 변형은 일반적이지만 가이드 RNA가 필요하지 않기 때문에 snoRNA가 뉴클레오티드 변형을 안내한다는 발견은 놀라운 일이었습니다.
그 후 수십 년 동안 수많은 snoRNA 유사 가이드 RNA의 기능이 밝혀졌습니다. 이제 그들은 스플라이싱에 관여하는 작은 핵 RNA(snRNA)와 어떤 경우에는 전달 RNA(tRNA)에서 부위 특이적 변형을 안내하는 것으로 알려져 있습니다. snoRNA의 하위 그룹은 또한 뉴클레오티드 변형을 넘어서는 기능을 나타내는 처리 전 rRNA의 구조적 재구성에 역할을 합니다. 또한 척추동물 텔로머라제에는 텔로미어 연장을 위한 주형 역할을 하는 snoRNA 유형 RNA 서브유닛인 TERC가 포함되어 있습니다. 진핵생물에서 아직 기능이 확인되지 않은 방대한 수의 snoRNA 유사 RNA를 고려할 때 향후 이러한 분자에 대한 새로운 역할이 더 많이 발견될 가능성이 높습니다.
진핵생물에서 아직 기능이 할당되지 않은 방대한 수의 snoRNA 유사 RNA를 고려할 때 향후 이러한 분자에 대한 더 많은 새로운 역할이 발견될 가능성이 높습니다.
가이드 RNA의 확장되는 우주
1990년대의 획기적인 발견에 이어 2000년대 초에는 광대한 새로운 작은 RNA의 세계가 발견되었습니다. 작은 간섭 RNA(siRNA), 마이크로RNA(miRNA) 및 PIWI 상호작용 RNA(piRNA)를 포함한 이러한 RNA는 가이드 RNA로 기능하여 단백질 복합체를 특정 RNA 표적으로 유도합니다(Bartel, 2004에서 검토됨; Czech, et al, 2018; 그림 1). siRNA는 내생적 센스 및 안티센스 전사체에서 형성되거나 바이러스 또는 합성 소스에서 외인성으로 도입될 수 있는 긴 이중 가닥 RNA(dsRNA)에서 유래합니다. miRNA와 piRNA는 서로 다르고 복잡한 성숙 경로를 통해 더 큰 전구체 RNA에서 처리됩니다. siRNA와 miRNA의 처리는 짧은 이중 가닥 RNA(dsRNA) 중간체를 생성합니다. 그 후, 표적 RNA를 보완하는 dsRNA의 가이드 가닥이 Argonaute 계열 단백질 구성원을 포함하는 단백질 복합체에 통합됩니다. 많은 Argonaute 단백질은 엔도뉴클레아제 활성을 가지고 있습니다. 표적과 완벽하게 상보적인 siRNA의 경우 Argonaute 단백질은 siRNA에 의해 지시된 부위에서 표적 RNA를 절단합니다. 종종 mRNA의 3′ 비번역 영역(3’UTR)을 표적으로 하는 miRNA에서 볼 수 있듯이 상보성이 불완전한 경우 결과는 번역 억제 또는 어떤 경우에는 번역 활성화입니다. piRNA는 이중 가닥 중간체를 형성하지 않지만 Argonaute 단백질의 특정 하위 계열과 결합합니다. 그들의 주요 기능은 전사 및 전사 후 메커니즘을 통해 생식 세포에서 전이인자 활성을 침묵시키는 것입니다.
본질적으로 이러한 작은 가이드 RNA 유사 분자는 유전자 조